最新发布第2页
文献阅读:脑膜对创伤性脑损伤和衰老的转录反应
摘要 研究人员利用高通量RNA测序(RNA-seq)技术,研究了脑膜对创伤性脑损伤(TBI)和衰老的转录反应,这一研究涵盖了亚急性和慢性的时间框架。研究人员首先使用单细胞RNA测序(scRNA-seq)探索...
Linux系统通过密钥ssh登陆配置方法
一、制作密钥对 1.打开windows或者Mac OS中的powershell,输入 #生成密钥对 ssh-keygen -t RSA -b 2048 -C "your@domain.com" #-t 后表示ssh的密钥类型,常用的有:rsa、ed25519、dss...
颅脑CT基础:CT断层解剖结构
一、颅CT基础 1.三种常用头颅扫描基线 (1)常规:横轴位(眦耳线),层厚8~10 mm (2)横轴位(眦耳线):上眶耳线(眦耳线向后倾斜20°) (3)鞍区扫描等 2.密度分类 低密度(黑-透光):空...
单细胞分析(一):矩阵数据的处理
一、总体代码 Seurat包官方文档https://satijalab.org/seurat/articles/pbmc3k_tutorial #加载所需R包 library(Seurat) library(tidyverse) library(dplyr) library(patchwork) library(ggplot2...
Hello, world!
疯狂肝了两天,此博客终于稳定下来。现在也终于可以有些时间写下第一篇文章,纪念一下这几天的付出。 一、建站灵感 说起建站的理由,其实也很偶然。前几天我在接收outlook邮件的时候,突然发现o...
文献学习2:创伤性脑损伤导致由小胶质细胞介导的慢性皮质炎症和神经元功能障碍
文献学习 Title:Traumatic Brain Injury Causes Chronic Cortical Inflammation and Neuronal Dysfunction Mediated by Microglia DOI: https://doi.org/10.1523/JNEUROSCI.2469-20.2020 题目...
WordPress跨服务器迁移最简单的手段
最近把博客建好了以后,一直在考虑如果有一天换服务器怎么办。于是就想把wordpress迁移流程走一遍,以便以后根据需要进行迁移。在这个过程中,又走了很多弯路,费了很多时间。也有一个好处,就...
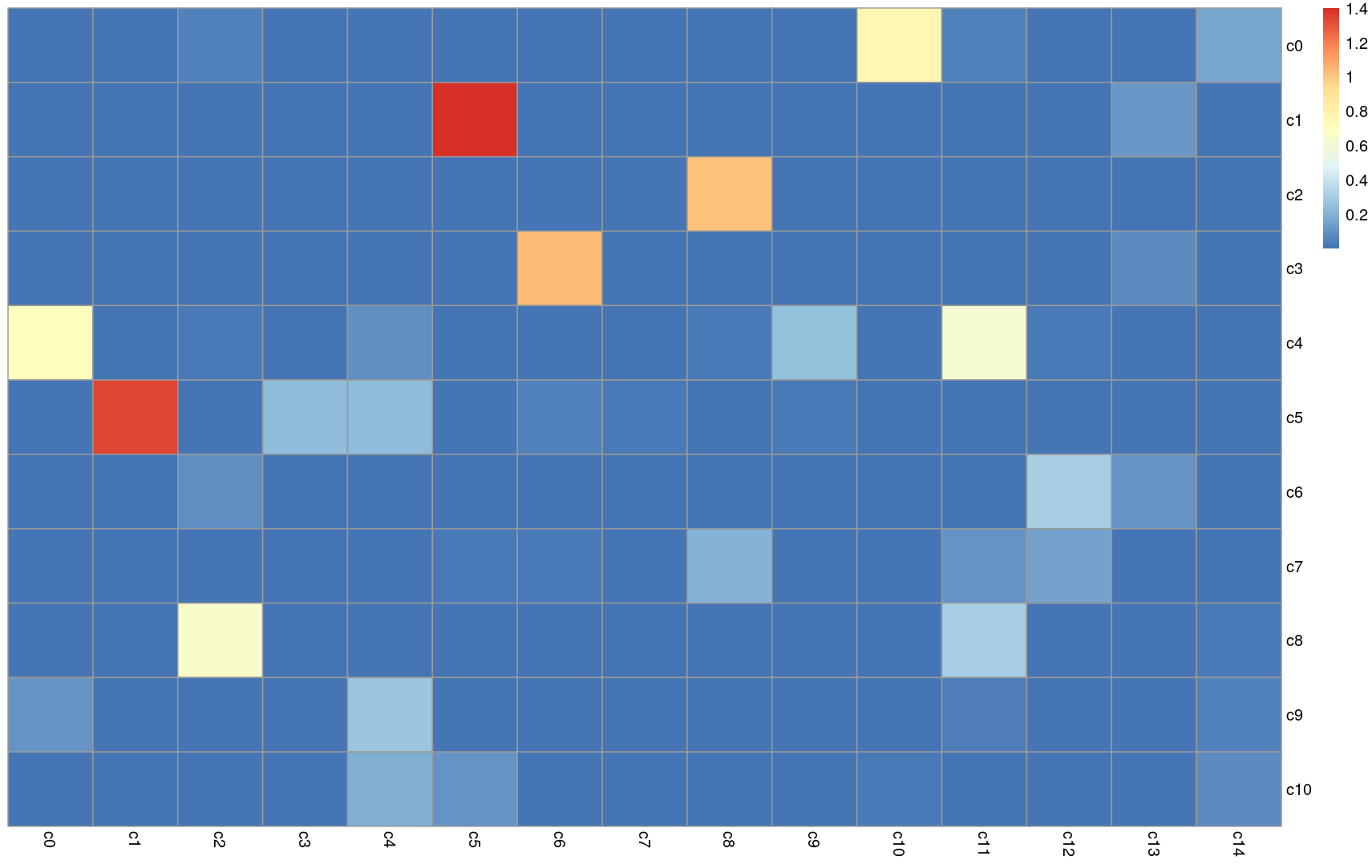
单细胞学习(五):通过聚类角度判断哪些cluster可能属于同一细胞类型
1.总体代码 ##清空环境 rm(list=ls()) #设置工作路径 ###加载所需要的包 library(Seurat) library(tidyverse) library(dplyr) library(patchwork) x=list.files() dir = c('BC2/', 'BC21/') nam...
小鼠中枢神经系统脱髓鞘和再髓鞘脑细胞转录组图谱和相互作用网络
code:https://github.com/marcocolonnalab/CPZ 原文地址:https://www.cell.com/cell-reports/fulltext/S2211-1247(23)00304-2?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2...
文献阅读:增强星形胶质细胞内的内源性大麻素信号可促进创伤性脑损伤的恢复
Title: Enhancing endocannabinoid signalling in astrocytes promotes recovery from traumatic brain injury DOI: https://doi.org/10.1093/brain/awab310 创伤性脑损伤是阿尔茨海默病和痴呆...